论文阅读_医疗知识图谱_GraphCare
1 | 英文名称: GraphCare: Enhancing Healthcare Predictions with Open-World Personalized Knowledge Graphs |
1 读后感
来自 230825 学习会 小丁分享
之前做医疗知识图谱和医疗预测时,最困难的问题包括:
- 如何结合现有的数据和知识
- 非结构化的文本类知识如何与数据结合
- 知识图结构如何设计,如何使用
- 如何引入时序的逻辑
- 如何使用大模型的知识和常识
不仅医疗领域,这些问题几乎存在于所有领域的建模。论文 GraphCare 对此进行了探索。这里只展示了不同知识图的优化效果,实际工作中,知识图生成的患者描述特征还可以与患者的检查,检验信息结合,使模型达到更优的效果。
2 介绍
文章主要针对的问题是:如何结合 患者情况 与 医疗知识 做出医疗预测。这里的医疗知识来自知识图谱,文章提出的改进主要针对知识图谱部分。包括以下三点:
- 使用大模型中的知识填充图谱中的知识盲区(题目中的 Open-world 概念)。
- 设计了针对个人的知识图谱结构。
- 提出了双向注意力增强 (BAT) 图神经网络 (GNN)。
数据使用 MIMIC 3/4,主要针对:死亡率、再入院、住院时间和药物建议 进行预测,与 GNN 相比几种预测的 AUROC 均有提升,且使用更少数据就能建模。
3 方法
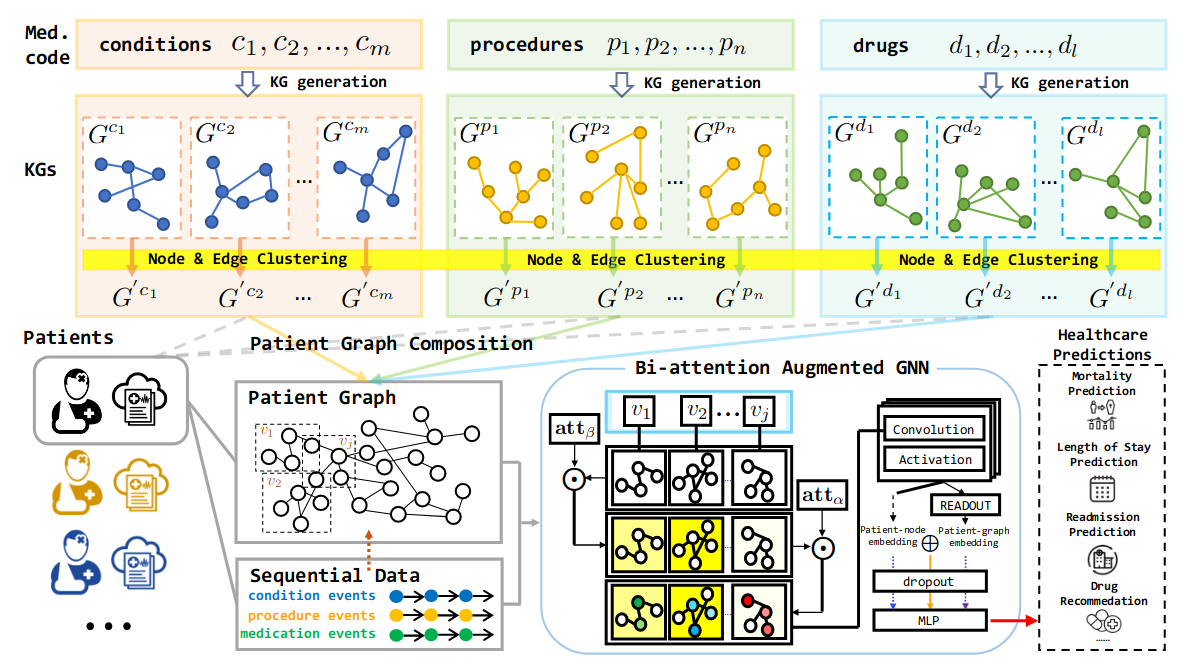
3.1 生成特定概念的知识图
这里的特定概念指的是医疗代码 e ∈ {c, p, d},三个字母分别代表:诊断、治疗和药物。对每个医疗代码,提取其知识图 Ge = (Ve, Ee),其中 V 是结点 E 是边。
3.1.1 建图
使用两种方法建图:
使用自然语言大模型构建
调用大模型,主要技术是设计提示模板,解析模型的返回结果,以及填充图。模板由三部分组成:指令(让模型做什么)、示例(返回结果什么样)、提示词(具体见附录 C.1);返回得到头实体,关系,尾实体的三元组;对于每个医疗代码,运行χ次,构造其知识图。
从现有知识图中取子图构建
为更好利用现有知识和知识图,通过子图采样提取医学代码的特定概念图。先识别现有生物医学知识图谱中与医学代码 e 对应的实体,然后随机采样源自实体的 κ 跳子图(具体见附录 C.2)。
3.1.2 对点和边聚类
使用文本构建的图,常会出现同概念对应多个描述的问题,这里使用聚类方法,合并概念。通过大语言模型,可以得到文本的嵌入,使用嵌入分别对结点和边聚类。
对全局图中相似的节点和边进行分组所有概念(如图 -1 所示)。经过聚类后,将原始图 G 中的节点 V 和边 E 映射到新的节点 V′ 和边 E′,获得新的全局图 G′ = (V′, E′),并为每个医疗代码创建一个新的图 G′e = (V′e, E′e) ⊂ G′。节点嵌入和边嵌入由每个簇中的平均词嵌入初始化。
4 生成患者知识图
这里分两个维度描述患者:患者医疗代码(得了什么病,不只一种病),患者的多次就诊。
针对每位患者,建立患者节点 P,并将其连接到图中的直接医疗代码。患者的个性化 KG 可以表示为 Gpat = (Vpat, Epat),其中 Vpat = P ∪ {V′e1, V′e2,..., V′eω };由于患者可能多次就诊,将患者 i 的访问子图可以表示为 Gpat(i) = {Gi,1, Gi,2,..., Gi,J } = {(Vi,1, Ei,1), (Vi,2, Ei,2),..., (Vi,J, Ei,J)}。
4.1 双向注意力增强图神经网络
图神经网络最终的输出一般是用向量 (数组) 表征的结点,比如最终用数组描述每位患者的情况,然后将患者作为实例,数组作为特征 X,以最终目标(如:是否死亡作)为 y 代入模型训练。以此实现对不同任务的预测。如果在训练模型时加入患者的其它特征,如实验室检验等数值型数据,模型就同时支持了患者数据和知识。
图神经网络的原理是聚合邻域节点信息来表示当前节点,从而学习图中的关系。相对于一般的 GNN 神经网络,文中提出了双向注意力增强网络(Bi-attention Augmented (BAT) GNN)机制。具体方法如下:
- 首先将词嵌入转换到隐藏嵌入减少节点和边嵌入的大小,以提高模型的效率并处理稀疏问题。
- 两个注意力权重:一个关注子图,一个关注子图中的结点:
\[ \begin{array}{l} \alpha_{i, j, 1}, \ldots, \alpha_{i, j, M}=\operatorname{Softmax}\left(\mathbf{W}_{\alpha} \mathbf{g}_{i, j}+\mathbf{b}_{\alpha}\right), \\ \beta_{i, 1}, \ldots, \beta_{i, N}=\lambda^{\top} \operatorname{Tanh}\left(\mathbf{w}_{\beta}^{\top} \mathbf{G}_{i}+\mathbf{b}_{\beta}\right), \quad \text { where } \quad \boldsymbol{\lambda}=\left[\lambda_{1}, \ldots, \lambda_{N}\right], \end{array} \]
患者 i 第 j 个访问子图中第 k 个节点的节点级注意力权重为 αi,j,k,患者 i 的第 j 次就诊,表示为 βi,j;g 描述 患者 i 第 j 次就诊是否涉及实体 k,M 是全局中的结点数,N 是最大就诊次数;W 和 b 是待学习的参数;λ是衰减系数,用于描述:就诊次数时间越接近,重要性越高。
参数的初始化利用了大模型返回的词嵌入,Wα的初值根据节点嵌入与目标(如死亡)的 cosine 距离设定,即节点描述与目标词义越相近,权重越高。最终计算出各个节点的隐藏层表示 h。
\[ \begin{array}{l} \mathbf{h}_{i}^{G_{\text {pat }}}=\operatorname{MEAN}\left(\sum_{j=1}^{J} \sum_{k=1}^{K_{j}} \mathbf{h}_{i, j, k}^{(L)}\right), \quad \mathbf{h}_{i}^{\mathcal{P}}=\operatorname{MEAN}\left(\sum_{j=1}^{J} \sum_{k=1}^{K_{j}} \mathbb{1}_{i, j, k}^{\Delta} \mathbf{h}_{i, j, k}^{(L)}\right), \\ \mathbf{z}_{i}^{\text {graph }}=\operatorname{MLP}\left(\mathbf{h}_{i}^{G_{\text {pat }}}\right), \quad \mathbf{z}_{i}^{\text {node }}=\operatorname{MLP}\left(\mathbf{h}_{i}^{\mathcal{P}}\right) \quad \mathbf{z}_{i}^{\text {joint }}=\operatorname{MLP}\left(\mathbf{h}_{i}^{G_{\text {pat }}} \oplus \mathbf{h}_{i}^{\mathcal{P}}\right), \end{array} \]
这里又针对每位患者计算 hiG 和 HiP,J 是就诊次数,K 是访问的节点数,1iΔ,j,k ∈ {0, 1} 是一个二进制标签,指示结点 vi,j,k 是否对应于患者 i 的直接医疗代码。我理解:前者是对与患者相关的所有节点取平均,后者是对与患者直接相关的医疗代码取平均。最终通过组合,使用 z 描述患者。
4.2 训练和预测
对于每位患者,考虑其 t 次就诊的数据:{(x1), (x1, x2),..., (x1, x2,..., xt)}
- 死亡率预测:利用患者前几次就诊预测未来是否死亡。
- 再入院预测:根据患者前几次住院情况预测患者 15 天以内的再入院。
- 住 ICU 时长预测:将问题定义为多分类,类别为: 1 天内,1 天,2 天... 7 天,一到两周,两周以上。根据本次及前几次就诊情况,判断住 ICU 时长。
- 推荐药物:根据本次及前次就诊,预测本次用药,用药可能为多种,因此定义为多标签问题。
5 实验
- EHR 数据,并使用公开的 MIMIC-III / MIMIC-IV 数据集。
- 构建知识图谱,使用 GPT-4 作为大模型,UMLS-KG 作为现有的大型生物医学知识图。
- 词嵌入,使用了 GPT-3 嵌入模型。
